更精确的RNA分子剪刀来啦!
RNA内切酶MazF —— RNA研究的新工具

小伙伴们已经看我们发了好多关于限制性内切酶的推文,有人会问了,你们天天在DNA上切来切去,有没有工具能够切RNA呢?这次我们就来向大家推荐一款可以在特定位置切割RNA的RNA内切酶——MazF。
No.1 MazF
MazF是细菌Type II“毒素-抗毒素 (toxin-antitoxin, TA)”系统”(听起来是不是跟“限制-修饰”系统很像呢)中的一种毒素蛋白 (toxin);它还有个形影不离的伙伴叫MazE,是一种抗毒素蛋白 (antitoxin)。平时MazF与MazE紧密结合,毒性被抑制。当收到外界胁迫等信号刺激时,MazF基因应激过表达,或者MazE蛋白降解,失去抑制的MazF蛋白开始切割mRNA,从而干扰(interfere)下游的一系列基因表达,产生细胞程序性死亡、细胞休眠等效应,从而应对外界胁迫。
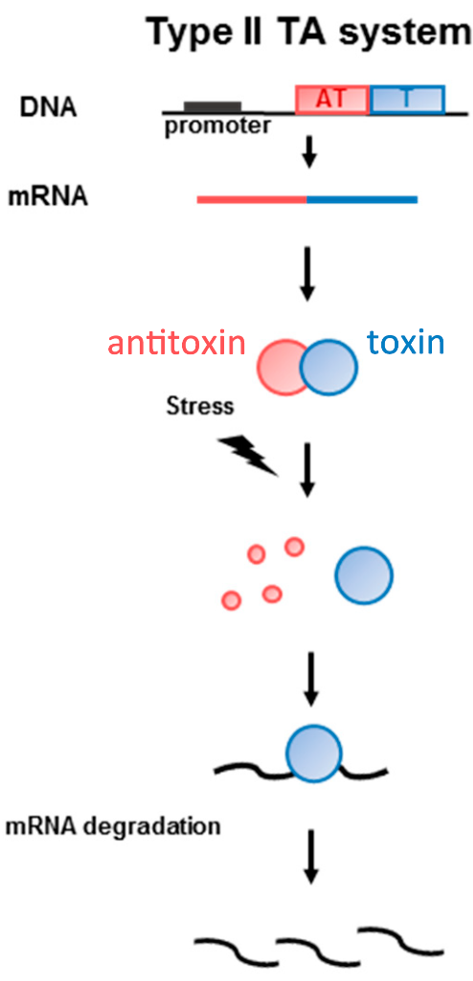
图1 Type II毒素-抗毒素系统
MazF是一个蛋白大家族,很多细菌中都含有这个家族的成员。最常见的MazF来源于大肠杆菌,特异性识别单链RNA中的ACA序列,最主要的切割位点是↓ACA,有时也会受上下游序列影响而变成A↓CA。
MazF对DNA和双链RNA都没有活性,如果RNA内部形成了二级结构,那么配对区域内的ACA就不能被切割。此外,MazF对m6A甲基化敏感,甲基化的m6ACA也不能被MazF切割。
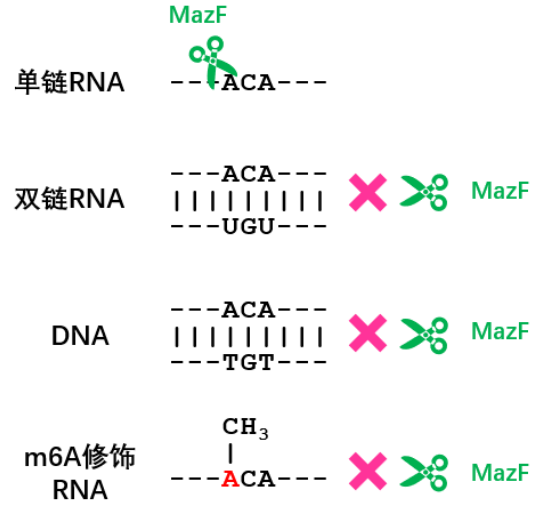
图2 MazF切割底物特征
MazF有两个性质与DNA限制酶存在显著差别:第一,MazF水解RNA磷酸二酯键的反应不依赖Mg2+,相反还会受Mg2+等二价金属离子抑制。第二,MazF切割RNA的磷酸二酯键之后,形成的两个末端分别是2’, 3’-环磷酸和5’-OH,产物不能直接用RNA连接酶重新连接。
No.2 MazF的应用
1. 高分辨率m6A甲基化分析
传统的高通量RNA m6A甲基化分析依赖于m6A抗体(如MeRIP-Seq),分辨率只能到100 – 200 nt,灵敏度、特异性、分辨率和样品量都存在较大限制。利用MazF不能切割m6ACA的性质,中山大学骆观正教授和以色列魏茨曼科学研究学院Schraga Schwartz教授在2019年分别独立开发了非抗体依赖的m6A高通量分析技术,各自命名为m6A-REF-seq和MAZTER-Seq。
两种技术采用相同的核心原理,都是用MazF来预处理mRNA,存在m6A修饰的ACA位点不会被切开,而没有甲基化的同样位点会被切开。然后将酶切后的片段建库测序,与参考序列比较,切开和未切开的位置,测序分析获得的丰度不一致,这样就可以在全转录组水平上,以单碱基分辨率量化分析m6A甲基化水平。
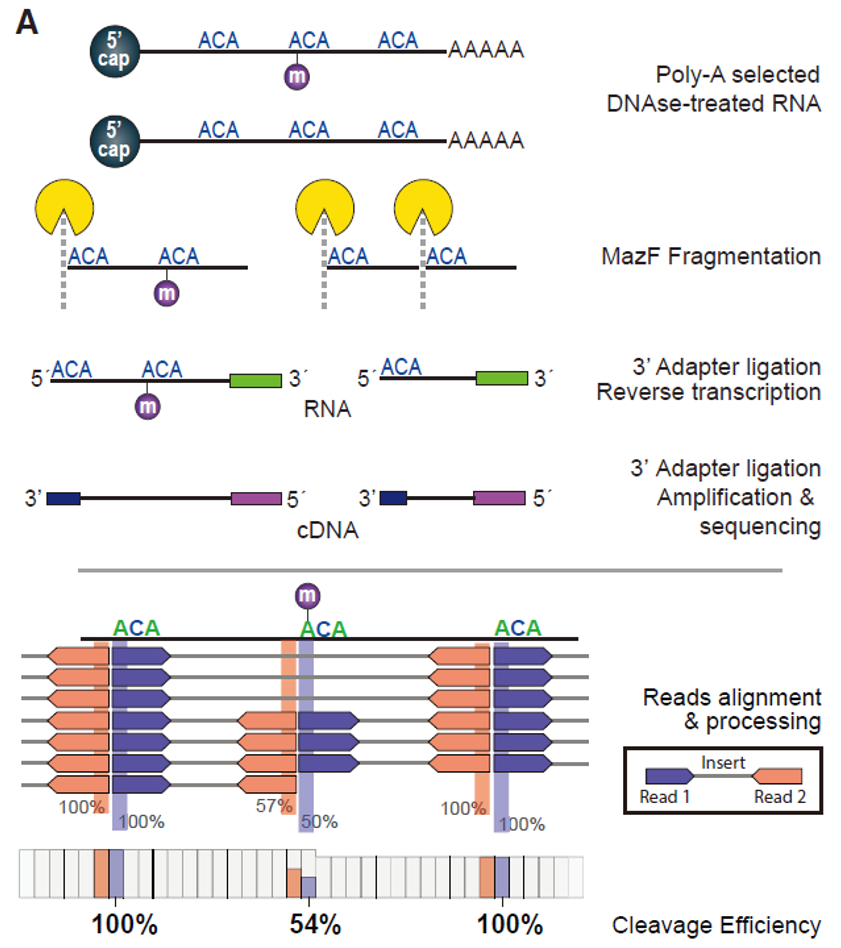
图3 MAZTER-Seq原理
此外,利用MazF和荧光定量PCR或基因芯片,也能够量化分析特定位点的m6A甲基化水平。
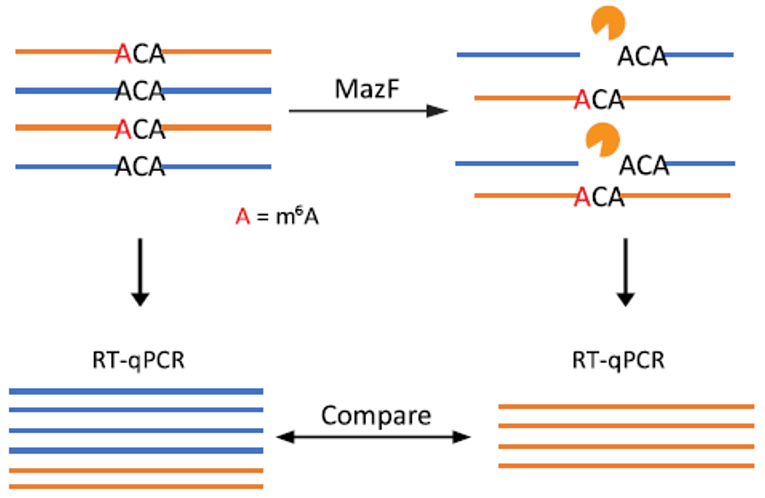
图4 m6A单碱基位点qPCR原理
2024年,复旦大学乔亮课题组又将MazF、滚环扩增 (RCA) 和基质辅助激光解吸电离飞行时间质谱 (MALDI-TOF MS) 结合起来,开发了一种新的检测m6A甲基化的方法。该方法可以在大量非甲基化RNA存在的情况下准确检测低含量的甲基化RNA,且检测限低,具有潜在的临床应用前景。
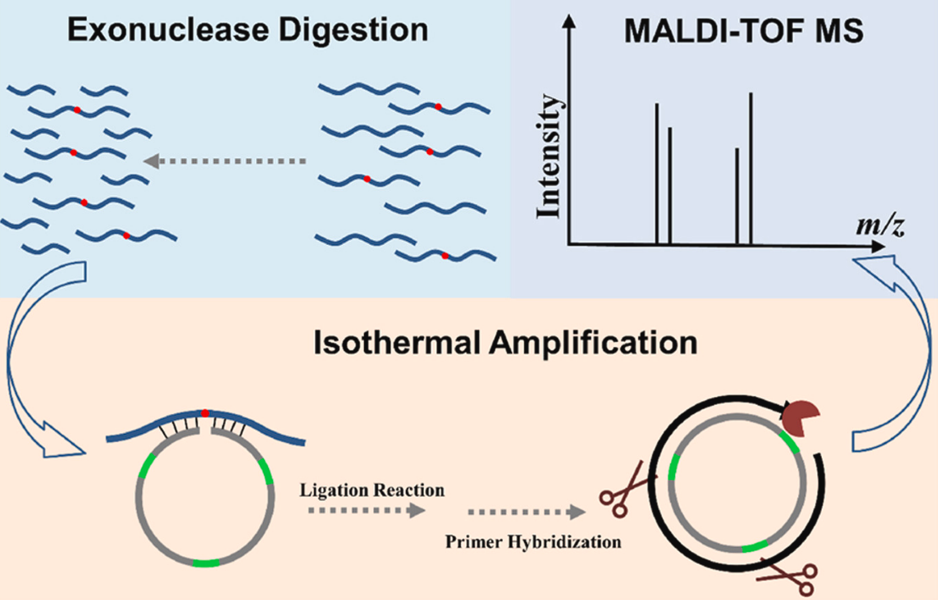
图5 MazF-RCA结合MALDI-TOF MS检测m6A甲基化原理
2. mRNA药物序列分析
与大多数生物药物一样,序列分析也是mRNA药物的一个关键质量属性。测序法虽然有效,但在生物制品的质量表征中,往往需要多种方法正交以获取更全面准确的信息。高分辨率质谱作为测序法的正交方法,具有独特的优势:无需扩增直接分析核酸(避免测序过程中的错配),更高的检测灵敏度,更短的分析时间,可直接检测修饰核苷酸等。
质谱分析mRNA序列的基本原理是,利用RNA酶将mRNA序列切割成短片段,然后用质谱分析每个片段的质量/电荷比,得到片段的精确分子量和二级碎片信息,从而获得准确的短片段序列和量化信息,再用“拼图”的策略将这些片段拼接起来得到完整mRNA的信息。
由于核酸仅有4个特定碱基,组合形式远小于蛋白质,因此需要酶解成较长的片段,以获得可以覆盖完整序列的特征片段。仅用特异性切割单链RNA上鸟嘌呤 (G) 的RNase T1,片段往往不能覆盖全序列。这时联合使用特异性更高的MazF等其他RNA内切酶,就能获得更好的mRNA mapping结果。
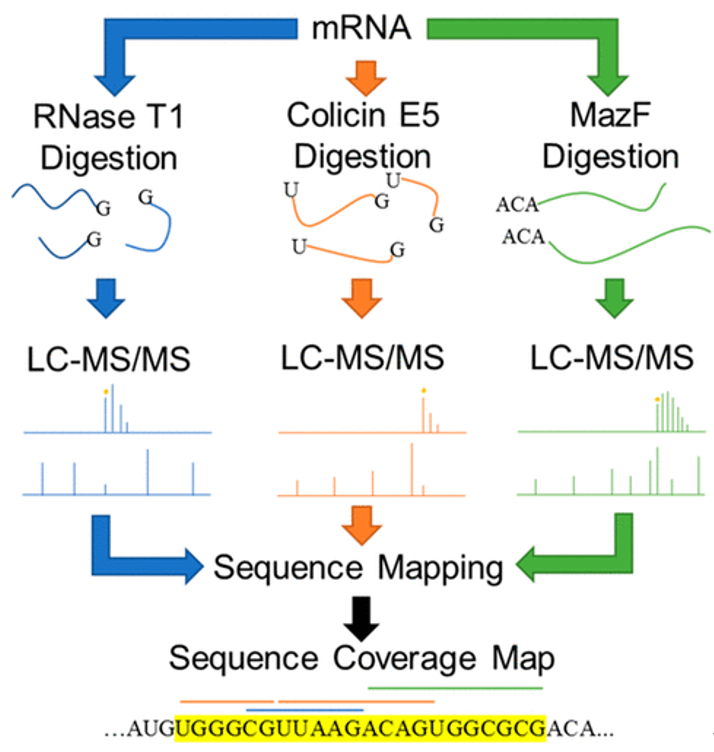
图6 多酶联合质谱分析mRNA序列原理
当然,与限制酶一样,仅有识别一种序列的RNA内切酶还远远不能满足需求。愚公生物将把RNA工具酶作为未来研发重点之一,为RNA工作者提供更多、更优质的工具。
相关产品

参考文献:
1. Mruk and Kobayashi. (2014) Nucleic Acids Res, 42:70-86
2. Culviner et al. (2018) Mol Cell, 70:868-880
3. Zhang et al. (2019) Sci Adv, 5: eaax0250
4. Garcia-Campos et al. (2019), Cell, 178:731–747
5. Han et al. (2024) Anal Chim Acta,1303:342532
6. Tao et al. (2019) Anal Chem, 91:8500–8506


